Il metodo
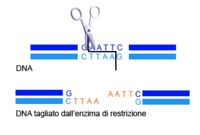
In natura gli enzimi di restrizione sono in larga parte ottenuti da batteri, i quali li utilizzano per tagliare il DNA dei virus che tentano di infettarli.
Esistono diversi enzimi di restrizioni, ciascuno specifico per una sequenza di DNA. I nomi degli enzimi di restrizione sono composti da tre lettere che derivano dall’iniziale del batterio da cui sono stati estratti la prima volta: per esempio, l’enzima Eco RI, deriva dal batterio E. coli. Gli enzimi di restrizione utilizzati per l’analisi dei VNTR hanno nomi come Hinf I, Hind III e Hae III.
In soggetti diversi questo tipo di taglio produce frammenti di DNA di lunghezza diversa, che può essere misurata mediante una tecnica chiamata elettroforesi su gel.
Il DNA è inserito in un supporto gelatinoso, posto all’interno di una cella elettroforetica a cui è applicata la corrente elettrica. Dal momento che il DNA è carico negativamente migra verso il polo positivo, con una velocità inversamente proporzionale alla dimensione: pertanto, bloccando la migrazione al momento opportuno, i frammenti saranno disposti dal più grande, vicino al punto di applicazione, al più piccolo, verso l’altro estremo del gel.
A questo punto si trasferisce il DNA dal gel ad una membrana più stabile, grazie ad un procedimento che sfrutta lo spostamento del DNA con i liquidi sottoposti a capillarità.
Si evidenzia la presenza dei frammenti (finora pressoché invisibili, o meglio non identificabili tra il resto delle molecole di DNA) con l’aiuto di molecole particolari chiamate sonde che si legano in modo specifico al DNA di interesse. Questo è possibile perché sono esse stesse fatte di DNA e presentano la stessa sequenza della porzione ricercata, che possono legare grazie alla natura stessa del DNA, molecola formata da due filamenti complementari, che possono essere separate e riassociate.
In questa procedura di analisi le eliche del DNA analizzate sono separate mediante una procedura chimica, esponendo in questo modo la faccia in grado di legare il filamento complementare. La sonda, agendo come filamento complementare, si lega al DNA sulla membrana.
Per rendere visibile questo legame si utilizzano delle molecole fluorescenti legate alle sonde. La membrana è messa a contatto con una pellicola fotografica che registra le emissioni luminose sotto forma di macchie. L’analisi delle macchie permette di identificare il tipo di porzioni di DNA presenti nel campione analizzato.
A seconda della specificità della sonda utilizzata si possono identificare delle variazioni minime nella sequenza (sonde ad alta specificità, riconoscimento di un singolo tipo di sequenza) o la presenza di determinati pattern di sequenza (sonde a bassa specificità, sonde che riconoscono molti tipi di sequenza).
Le sonde ad alta specificità sono utilizzate principalmente per l’analisi di paternità.




